La vida es algo muy complicado. Los mecanismos que mantienen una sola célula funcionando son ya de por sí, difíciles de entender. En organismos multicelulares, la dificultad aumenta exponencialmente. ¿Crees que hemos llegado al punto de poder controlar y crear vida en un laboratorio? La realidad es que la biotecnología y la biología sintética nos permiten acercarnos a esta idea, pero, ¿Podemos jugar a ser dioses? Hoy voy a hablarte de lo cerca que estamos de crear vida en un laboratorio.
Índice de contenido:
¿Qué es la vida?
Existen muchas definiciones de vida, algunas más restrictivas que otras. Tradicionalmente, consideramos vivo aquel ser que, de manera autónoma, puede reproducirse, dejar su información como legado y mantenerse, generando energía a través del metabolismo. Otras definiciones simplifican el concepto, definiendo ser vivo como cualquier ente que pueda reproducirse, mutar y seguir las leyes de la selección natural.
Según esta última definición, los virus podrían ser seres vivos aunque no sean más que un poco de material genético cubierto por unas cuantas proteínas. Pueden mutar y la selección natural puede favorecer estos cambios. No obstante, casi todos los expertos niegan que los virus estén vivos, ya que para replicarse necesitan la maquinaria de otra célula. De modo que, quizás, crear vida sea mucho más fácil de lo que parece. Según las definiciones más transigentes, para crear vida sólo necesitaríamos un poco de material genético, una envoltura que lo proteja y las proteínas necesarias para replicarse. ¿Lo hemos conseguido?
Bacterias sintéticas
En 2006 se patentó el genoma de la bacteria Mycoplasma laboratorium. Este incluye lo justo y necesario para mantener viva una célula capaz de autorreplicarse, junto con unas secuencias denominadas «marcas de agua» que permiten identificar el genoma con facilidad. ¿Es esto crear vida? El grupo de Craig Venter, que patentó esta hazaña, publicó en 2010 un artículo en Science que describe el proceso de síntesis de un organismo sintético, utilizando Mycobacterium mycoides, una bacteria similar.
No obstante, M. laboratorium no fue el primer ser vivo sintetizado químicamente. En el 2003 ya se había conseguido recrear una estructura vírica, concretamente del fago φX174. Este hecho no tuvo la misma importancia que la bacteria sintética para la comunidad científica. El reto de sintetizar un virus fue mucho menor, ya que su genoma es 50 veces más pequeño. Esto allanó las bases para crear vida bacteriana en un laboratorio.
Sintetizar un genoma
Desde hace años es posible sintetizar ADN de manera química, pero, ¿Cómo es posible crear un genoma de millones de nucleótidos?
El grupo de Gibson y Venter consiguió sintetizar químicamente fragmentos de 1080 nucleótidos. Gracias a la levadura Saccharomyces cerevisiae, consiguieron unir grupos de 10 fragmentos por homología, generando piezas de unos 10.000 nucleótidos. Repitiendo el proceso varias veces, consiguieron generar un genoma completo y circular, en el que el último fragmento se uniese con el primero.
Así, fue posible ensamblar el genoma completo de Mycobacterium. Más de un millón de nucleótidos, todos sintetizados químicamente, con la información básica que aseguraba la supervivencia de la célula. Junto a los genes para la replicación, metabolismo y de respuesta a estímulos nativos, se incluyeron marcas de agua (para reconocer las secuencias como propias) y pequeños genes, como el LACZ, que permitían reconocer visualmente la nueva cepa.
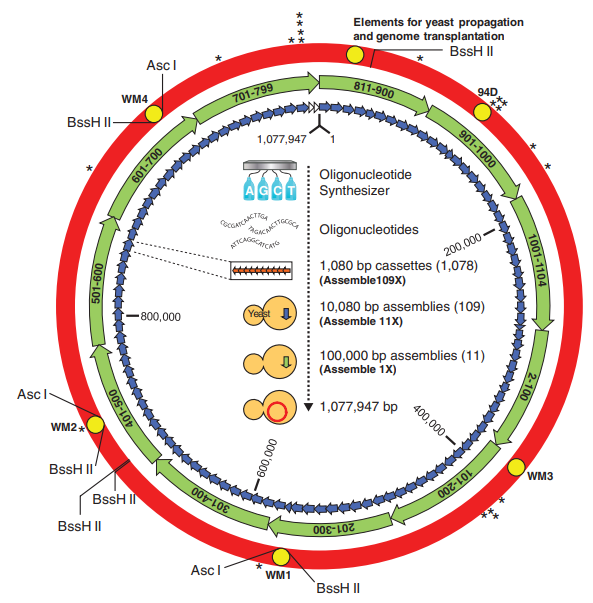
Trasplante del genoma
Aún conseguido el genoma, falta todo lo demás. Se extrajo el genoma sintético de la levadura que lo contenía y se inyectó en una célula bacteriana desprovista de él (es decir, se hizo un trasplante).
El resultado fue una cepa sintética de Mycobacterium mycoides. Fácil de reconocer, no sólo por las marcas de agua en el genoma sintético, sino también porque fenotípicamente era muy diferente a las cepas silvestres.
¿Y crear vida eucariota?: Proyecto Syntetic yeast 2.0
El proyecto «Sc2.0» tiene como objetivo sintetizar el genoma de la levadura Saccharomyces cerevisiae. Esto supone todo un reto, ya que cuando hablamos de «crear vida» no es lo mismo una bacteria que un organismo eucariota, por sencillo que sea. Los orgánulos suponen un problema, hay un núcleo, con un genoma es mucho mayor, organizado en cromosomas y no circular.
Este proyecto ha involucrado durante años a diversos laboratorios de todo el mundo bajo el liderazgo de Jef Boeke. Saccharomyces cerevisiae tiene 16 cromosomas, y cada grupo colaborador participa en la síntesis y ensamblaje de uno (o parte) de ellos.
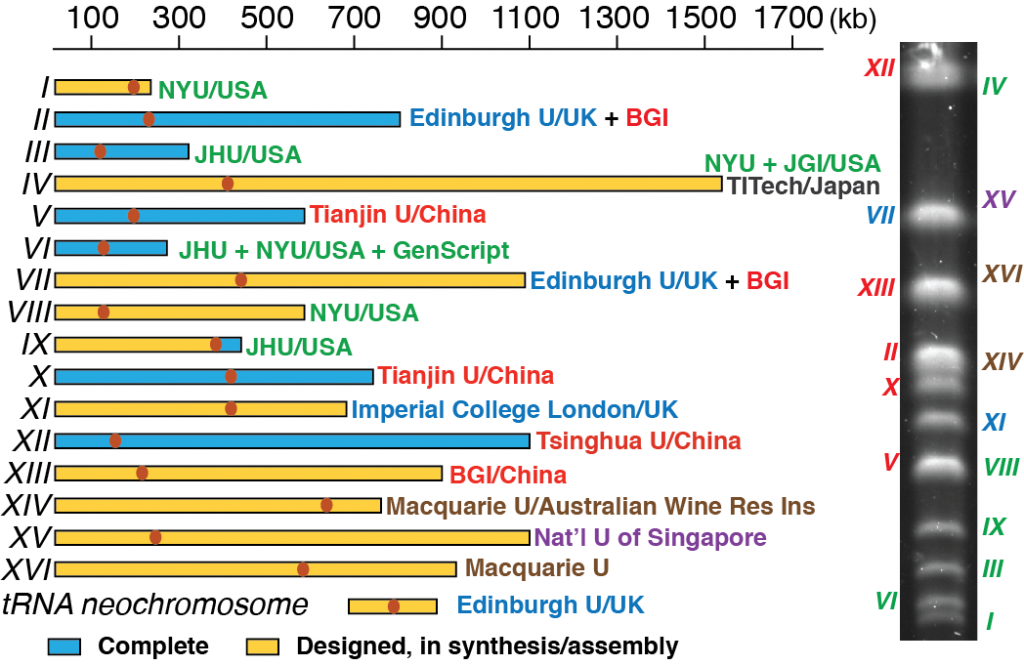
Aunque el genoma de esta levadura sintética no está todavía finalizado, se puede comprobar el avance y el estado de síntesis de cada uno de los cromosomas, que serán versiones ligeramente distintas a los originales (sin las secuencias no indispensables y con marcas de agua).
El objetivo no es solo crear vida a través de una cepa de levadura sintética. También se busca lograr una especie optimizada, perfecta para trabajar con ella en el laboratorio. Por ello han insertado en los nuevos cromosomas un sistema denominado SCRaMbLE, por sus siglas «Recombinación y modificación cromosómica sintética mediante evolución activada por LoxP». Con este sistema se busca inducir modificaciones genómicas. Mutaciones, o incluso traslocaciones de fragmentos grandes de ADN serían inducibles a voluntad con este sistema, que pretende versatilizar y optimizar el manejo de Saccharomyces. De este modo, podremos jugar a inducir eventos evolutivos… O, como algunos otros dirían, «a ser dioses».

¿En qué punto estamos?
Crear vida en un organismo a partir de cero no es tarea sencilla, pero es posible. Con todo el conocimiento adquirido a lo largo de los años se ha visto que el mayor problema es crear un genoma estable. En el mejor de los casos, se ha logrado tan solo crear y trasplantar un genoma de una bacteria a un «recipiente» que ya estaba vivo. Actualmente se está trabajando en originar una célula completa, en la que tanto la membrana plasmática como las proteínas y el material genético sean sintéticos.
El objetivo real es conseguir células programables, óptimas para su uso en un laboratorio y que no puedan sobrevivir en el exterior, para no perturbar el medio ambiente. Aunque la tecnología existe, está claro que lo que hacemos con ella es lo que define nuestras intenciones. De modo que, respondiendo a la pregunta que encabeza este artículo, no, no podemos jugar a ser dioses. Nuestra ética como científicos nos impide crear vida por el simple placer de hacerlo. Debe haber siempre una intención de mejora, de optimización. Y mucho, mucho cuidado.
Artículos que pueden interesarte
- ¿Cómo se descubre la causa genética de una patología?
- El genoma y los mecanismos epigenéticos
- Las mutaciones silenciosas: ¿Cómo se generan las proteínas y qué efecto pueden tener sobre ellas?
- No puedo vivir sin ti: los genes esenciales para vivir.
Referencias
- McKay, C. P. 2004. What Is Life – and How Do We Search for It in Other Worlds? Plos Biol, 2(9): e302. doi: 10.1089/ast.2010.0524.
- Hamilton O. Smith, Clyde A. Hutchison III, Cynthia Pfannkoch, and J. Craig Venter. 2003. Generating a synthetic genome by whole genome assembly: φX174 bacteriophage from synthetic oligonucleotides. PNAS, 100 (26): 15440-15445. doi: https://doi.org/10.1073/pnas.2237126100.
- Gibson, D. G., Glass, J. I., Largitue, C:, Noskov, V. N., … Venter, C.J. 2010. Creation of a Bacterial cell controlled by a chemically synthesized genome. Science, 329 (5987): 52-56. doi: 10.1126/science.1190719.
- Steensels, J., Gorkovskiy, A., Verstrepen, K. J. 2018. SCRaMbLEing to understand and exploit structural variation in genomes. Nature com. 9 (1937) doi: https://doi.org/10.1038/s41467-018-04308-3.







