Durante más de dos años la palabra «virus» ha formado parte de forma rutinaria de la mayoría de las conversaciones, y es que la COVID-19 ha dado un giro de 360º a nuestras vidas. Como hemos podido comprobar, un virus de menos de una micra ha conseguido paralizar el planeta entero. Sin embargo, ¿qué ha pasado con el resto de enfermedades y de microorganismos? ¿qué son las bacterias ESKAPE? ¿Han pasado a un segundo plano debido a la importancia actual del SARS-CoV-2?
En este artículo relato el gran problema que resultan las bacterias multi-resistentes a los antibióticos. ¿Estamos a tiempo de solucionar el problema que originan las mismas, o nos encontramos ante el comienzo de una nueva pandemia? Si tienes interés por este tema, acompáñame a averiguar qué ocurre en la actualidad con las bacterias y su resistencia a los antibióticos.
Índice de contenido:
Introducción
Antibióticos
Desde su introducción clínica, los antibióticos han influido enormemente en la vida en la Tierra, al ser la principal solución a las infecciones bacterianas. En los últimos 80 años, los antibióticos han propiciado grandes avances en la medicina moderna y han revolucionado las prácticas agrícolas e industriales. Además, cabe destacar que el uso generalizado de los antibióticos se asemeja a la ubicuidad de las propias bacterias.
Los antibióticos son agentes antimicrobianos producidos por los microorganismos (antibióticos naturales). Aunque se conocen miles de antibióticos, menos del 1% son clínicamente útiles, debido principalmente a problemas de toxicidad o la falta de absorción por las células del hospedador. Los antibióticos naturales que son modificados artificialmente para mejorar su eficacia son los antibióticos semisintéticos.
Son muchas las clases de antibióticos existentes, y perfectamente se podría escribir un artículo exclusivamente sobre ellos. Simplificando, los antibióticos que suponen más de la mitad de todos los antibióticos producidos y utilizados en el mundo son los β-lactámicos, que son inhibidores de la síntesis de la pared celular de las bacterias. Dentro de los ellos se encuentran numerosos ejemplos de gran importancia clínica, como la penicilina, las cefalosporinas o los carbapenémicos.
Otros grupos de antibióticos que también presentan importancia son los aminoglucósidos (ej., estreptomicina, kanamicina), macrólidos (ej., eritromicia, azitromicina), tetraciclinas (ej., doxiciclina), quinolonas (ej., levofloxacino ) o glucopéptidos (ej., vancomicina), entre muchos otros.
Resistencia a los antibióticos
Los antibióticos han facilitado avances tanto en los viajes como en el comercio mundial, pero a su vez han contribuido a la rápida difusión de la resistencia antimicrobiana (AMR).
La AMR es un fenómeno microbiológico natural. En su lucha por la supervivencia en la Tierra primitiva, las bacterias se vieron obligadas a desarrollar mecanismos para establecer y defender sus nichos ecológicos. A lo largo de miles de millones de años, las bacterias han desarrollado mecanismos para evitar, expulsar, negar, destruir o resistir compuestos estructuralmente similares a los antibióticos de uso clínico en la actualidad. Estos mecanismos fisiológicos intrínsecos constituyen la base evolutiva de la resistencia a los antibióticos antropogénicos.
Para más información acerca de los mecanismos que presentan las bacterias para sobrevivir a los antibióticos, podéis leer el artículo de Alejandro Lucas, donde habla con más detalle de los mismos.
Las advertencias sobre la resistencia precedieron al uso clínico de los antibióticos y, desde entonces, la introducción de cada nuevo antimicrobiano ha sido seguida de cerca por la aparición de resistencia contra él. El uso exorbitante de los antibióticos ha ejercido una presión selectiva hacia la AMR de alto nivel y la resistencia a múltiples fármacos, haciendo que clases enteras de antibióticos sean redundantes y amenazando con provocar el fin de la «era de los antibióticos».
El problema de la aparición de bacterias resistentes a los antibióticos es cada vez más real, notorio y preocupante. Asimismo, en los últimos años se ha relacionado la aparición de cepas bacterianas multirresistentes a los antibióticos (bacterias que son resistentes a más de tres clases de antibióticos) con la disminución del desarrollo de nuevos antibióticos.
Por ello, la Organización Mundial de la Salud (OMS) y los Centros para el Control y Prevención de Enfermedades de Estados Unidos (CDC) establecieron que las bacterias resistentes a los antimicrobianos son una amenaza inminente. Tal afirmación es respaldada por cifras: por ejemplo, en Europa, cada año más de 33.000 muertes se atribuyen a las infecciones originadas por las bacterias resistentes a los antibióticos adquiridas en el hospital y en la comunidad, lo que supone elevados costes directos e indirectos a la sociedad.
Cabe destacar que la escasez de métodos de rápido diagnóstico para la identificación de los patógenos bacterianos y de los genes de resistencia a los antibióticos en los entornos clínicos, ha dado lugar al uso reiterado e innecesario de antibióticos de amplio espectro de actuación (antibióticos que no están dirigidos exclusivamente a las bacterias causantes de la infección).
Un término de gran importancia dentro del ámbito de los antibióticos y de las bacterias resistentes a los mismos es el ST o concepto de clon. Para entender este concepto, en primer lugar hay que saber cuál es la composición del genoma bacteriano (Fig. 1).
- Este está compuesto por un conjunto de genes que forman el genoma core, que son los genes conservados en todos los aislados de una especie.
- El resto de genes conforma el genoma variable o accesorio, que es diferente entre los diferentes aislados de una especie.
Para poder anotar y estudiar el genoma de una especie se han elegido entre 7-10 genes que están muy conservados (no sometidos a presión selectiva) dentro de una especie, para definir el secuenciotipo (ST) o clon. Por tanto, para establecer un nuevo ST o clon se estudian las variantes alélicas de dichos 7-10 genes conservados. De manera que cada vez que se identifica una variante alélica diferente en uno de los genes conservados, se define un nuevo ST de la especie que se está estudiando.

Definición y bacterias a las que engloba
En definitiva, en un intento por centrar la atención y orientar la investigación y el desarrollo relacionados con nuevos antibióticos, la OMS publicó en febrero de 2017 un listado de patógenos para los que se necesita urgentemente el desarrollo de nuevos antimicrobianos. Dentro del gran listado, los patógenos conocidos como ESKAPE (Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa y especies de Enterobacter), fueron designados como prioritarios en la búsqueda de nuevos fármacos.
Los patógenos presentes en el grupo ESKAPE, como su propio nombre indica, son capaces de “escapar” a la acción letal de los antibióticos.
Un mayor conocimiento de la virulencia, la resistencia, la transmisión y la patogenicidad de estos microorganismos puede conducir a estrategias innovadoras para el desarrollo de nuevas opciones antimicrobianas. Además, el hecho de prestar una mayor atención a los microorganismos más patógenos ayudará a centrar el desafío de la AMR y permitirá una eficaz evaluación de los nuevos agentes antimicrobianos.
Descripción de las bacterias ESKAPE
Enterococcus faecium (E):

Enterococcus faecium es un patógeno Gram-positivo, anaerobio y oportunista, frecuentemente implicado en las infecciones asociadas a la atención sanitaria, especialmente entre los pacientes inmunodeprimidos (Fig. 2). En el entorno hospitalario, los enterococos se han descrito como patógenos de «triple amenaza» que presentan una colonización importante del intestino y la piel junto con una importante persistencia en el medio ambiente.
La resistencia a los antibióticos β-lactámicos está garantizada en E. faecium, con la prevalencia de la resistencia completa a ampicilina e imipenem. Además, las cepas de esta especie están presentando una mayor resistencia a la vancomicina (antibiótico dentro de la clase glucopéptidos). Además, el clon ST17 de E. faecium resistentes a la vancomicina (VREfm) es actualmente responsable de un importante número de infecciones hospitalarias.
En comparación con la duración de los brotes causados por los otros patógenos del grupo ESKAPE, como más adelante se verá, los brotes de VREfm tienen una larga duración, de aproximadamente 11 meses. La entrada de las bacterias VREfm en el torrente sanguíneo de los pacientes hospitalizados suele ir precedida de una exposición a los antibióticos. Además, el principal problema es la facilidad que presentan para la formación de biopelículas o biofilms.
El tratamiento de la infección se basa en terapias antibióticas de segunda línea, que se suelen asociar con un mayor coste, una menor eficacia y un mayor riesgo de toxicidad.
Staphylococcus aureus (S):
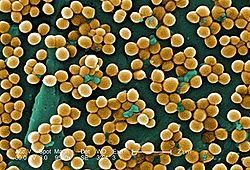
Staphylococcus aureus es un representante común de la microbiota de la piel y se aísla con mayor frecuencia de zonas húmedas, como las fosas nasales y las axilas (Fig. 3). Alrededor del 60% de la población humana alberga S. aureus de forma irregular, mientras que alrededor del 20% de los individuos son portadores persistentes (casi siempre portadores de una única cepa de S. aureus).
Esta bacteria es un destacado patógeno de las heridas, capaz de provocar infecciones tanto agudas como crónicas tras la formación de un biofilm (una de las principales causas de infección crónica tras la inserción de dispositivos médicos implantados, como válvulas cardíacas mecánicas o dispositivos ortopédicos).
Una de las cepas más importantes dentro de S. aureus es SARM, que presenta resistencia a los antibióticos β-lactámicos. Estas cepas pueden llegar a provocar infecciones de la piel y de los tejidos blandos, infecciones en el torrente sanguíneo (suponen el 20% de la mortalidad originada por esta bacteria) o neumonías graves. Entre los diferentes clones, el SARM ST80 es uno de los principales causantes de infecciones en entornos sanitarios.
El tratamiento de primera línea para las infecciones por SARM en todo el mundo son los antibióticos glucopéptidos, como la vancomicina o la teicoplanina. Sin embargo, esta intensa presión selectiva ha provocado la previsible aparición de S. aureus con resistencia intermedia a la vancomicina (VISA; prácticamente insensible a todos los antibióticos, salvo los más reservados) y resistente a la vancomicina (VRSA). Finalmente, destacar que el S. aureus resistente a la oxacilina (BORSA) no son ni resistentes ni sensibles a la meticilina y su identificación suele ser errónea, lo que amenaza el tratamiento de los pacientes.
Klebsiella pneumoniae (K):

Klebsiella pneumoniae es un miembro destacado de la familia de las enterobacterias Gram-negativas (Fig. 4), junto con Escherichia coli, y juntas lideran una creciente amenaza patológica. Son capaces de provocar una infección invasiva y han adquirido una gran variedad de enzimas β-lactamasas capaces de hidrolizar el anillo β-lactámico común en penicilinas, cefalosporinas e incluso carbapenémicos.
Los antibióticos de tipo cefalosporina y los carbapenémicos han sido un pilar fundamental en el tratamiento de las infecciones graves causadas por Enterobacterias como K. pneumoniae (mortalidad superior al 40%), pero su eficacia se ha visto comprometida por la adquisición generalizada de genes que codifican otros tipos de enzimas que provocan resistencias, como las BLEEs, las carbapenemasas o las NDM. Por tanto, se carece de antibióticos eficaces, y el tratamiento suele requerir la utilización de fármacos con riesgo de toxicidad.
Las cepas de K. pneumoniae resistentes a los carbapenémicos (CRKP) son las enterobacterias resistentes a los carbapenémicos más destacadas desde el punto de vista clínico. La diseminación global de la CRKP queda ejemplificada por el clon ST307, que se ha diseminado con éxito por todos los continentes.
Acinetobacter baumannii (A):
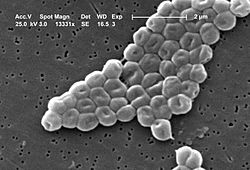
Acinetobacter baumannii es un patógeno oportunista que se encuentra con mayor frecuencia en las UCI y en las salas de cirugía, donde el uso prolongado de antibióticos ha permitido la selección de resistencia a todos los antimicrobianos conocidos (Fig. 5).
Al igual que otros Gram-negativos, A. baumannii es conocido por su persistencia en el medio ambiente. Presenta mayor resistencia a las condiciones de sequedad y puede crecer en un rango de temperaturas, pHs y niveles de nutrientes muy variado, lo que hace que esté altamente adaptado a la supervivencia en vectores humanos o ambientales.
Es intrínsecamente resistente a los antibióticos y las infecciones causadas suelen producirse en pacientes hospitalizados. Aunque las tasas de infección por A. baumannii son comparativamente bajas en comparación con las de otros patógenos de la clasificación ESKAPE, aproximadamente el 45% de todos los aislados de A. baumannii en el mundo se consideran multirresistentes, por lo que los antibióticos de último recurso de tipo carbapenémico y polimixina ya no son eficaces.
En definitiva, esta combinación de virulencia intrínseca y múltiples factores de resistencia convierte a A. baumannii en un paradigma de patogenicidad con pocos o ningún compuesto candidato en fase de desarrollo para el tratamiento de las infecciones causadas. Si no se toman medidas adecuadas mediante la mejora de la vigilancia epidemiológica y el desarrollo terapéutico, A. baumannii tiene la capacidad de provocar una epidemia mundial.
Pseudomonas aeruginosa (P):

Pseudomonas aeruginosa es una bacteria Gram-negativa, anaerobia facultativa, que sobrevive en condiciones con poco oxígeno (Fig. 6). Es capaz de sobrevivir en entornos extremos (forma biofilms) y se encuentra muy presente en el entorno hosìtalario, por lo que es una de las principales causas de infecciones (es responsable del 10% de todas las infecciones nosocomiales).
P. aeruginosa coloniza preferentemente a los pacientes inmunocomprometidos y, como tal, se conoce como un patógeno oportunista. La adherencia inicial dentro de un huésped se ve favorecida por el uso de un flagelo y los pilli. Una vez adherida, P. aeruginosa emplea sus pigmentos para superar la falta de hierro e imponerse a las respuestas inmunitarias innatas.
P. aeruginosa es inherentemente resistente a una gran variedad de antimicrobianos, como las fluoroquinolonas. Además, la adaptabilidad del genoma de P. aeruginosa es clave en la capacidad del patógeno para persistir crónicamente en el huésped y evadir el tratamiento antibiótico. Cabe destacar que son los clones ST235 y ST175 de P. aeruginosa los que mayor dispersión presentan y son principales responsables de las infecciones hospitalarias.
Además de las mutaciones puntuales que le confieren la resistencia, se sabe que P. aeruginosa expresa β-lactamasas de amplio espectro, lo que da lugar a una gran resistencia a los carbapenémicos. Hay pocos o ningún fármaco en fase de desarrollo clínico para hacer frente a este patógeno.
Especies de Enterobacter (E):
Las especies de Enterobacter suelen infectar el tracto urinario y respiratorio, pero se sabe que también causan infecciones del torrente sanguíneo e infecciones nosocomiales (Fig. 7). Muestran una amplia resistencia a los antibióticos debido a la presencia de BLEEs. En los últimos 35 años, las especies de Enterobacter aerogenes (actualmente renombrada como Klebsiella aerogenes) y Enterobacter cloacae se han presentado como amenazas significativas para las salas de neonatos y los pacientes de las UCI.
Las especies de Enterobacter MDR son una causa creciente de infecciones hospitalarias. En el caso del complejo E. cloacae, la resistencia a los carbapenémicos se ha extendido debido a la diseminación de los clones ST178 y ST78 resistentes a los carbapenémicos. Esto es preocupante, ya que antes de 2005 aproximadamente el 99,9% de las cepas de Enterobacter eran sensibles a los carbapenémicos.
Para complicar aún más el tratamiento de las infecciones bacterianas, E. aerogenes es capaz de albergar subpoblaciones de bacterias resistentes a la colistina que son indetectables mediante las actuales pruebas de diagnóstico. Aparte de la colistina, pocos antimicrobianos son eficaces contra estas bacterias y, al igual que con otros patógenos ESKAPE, hay pocos o ningún fármaco en desarrollo que se sepa que sea capaz de hacer frente a esta creciente crisis sanitaria.
Formas de «eskapar» de los antibióticos de las bacterias ESKAPE
Dada la frecuencia con la que se encuentran los organismos ESKAPE en el entorno clínico, no es de extrañar que se observen numerosos mecanismos diferentes de resistencia a los antibióticos en estos patógenos, como bien se ha ido mencionando. Éstos pueden clasificarse a grandes rasgos en cuatro grupos (Fig. 8), que comprenden:
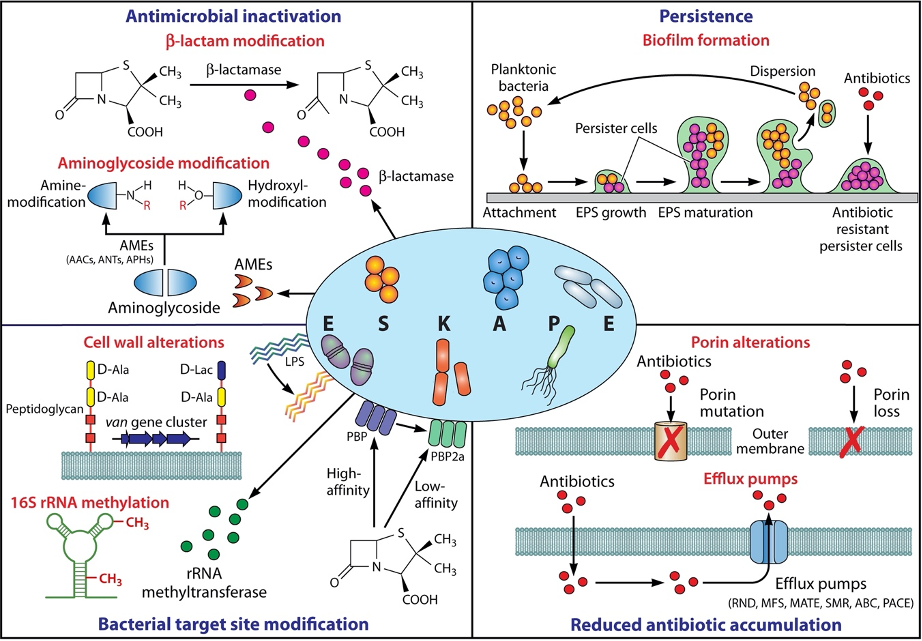
- La inactivación o alteración de la molécula antimicrobiana:
Es uno de los mecanismos de AMR más comúnmente empleados por los patógenos ESKAPE. Implica la producción de enzimas que destruyen o neutralizan irreversiblemente los antibióticos. Dichas enzimas son especialmente frecuentes entre los patógenos Gram-negativos y comprenden aquellas que destruyen el sitio activo del antibiótico o que modifican elementos clave del fármaco.
Un ejemplo claro son las β-lactamasas , que se identificaron por primera vez poco después de la purificación de la penicilina. Desde entonces, se han descrito unas 2.600 β-lactamasas únicas que permiten la resistencia a uno o más antibióticos β-lactámicos. Son el mecanismo de resistencia más importante entre los patógenos ESKAPE. Se localizan dentro del periplasma de la bacteria, hidrolizando así los β-lactámicos antes de que estos alcancen su sitio diana.
- Las modificaciones del sitio diana bacteriano:
Esta estrategia reduce la afinidad o impide la unión del antibiótico al sitio diana. En concreto, estos mecanismos incluyen la modificación de la enzima diana, las alteraciones del sitio diana ribosomal y las alteraciones del precursor de la pared celular.
- La reducción de la penetración/acumulación de antibióticos:
Dentro de este mecanismo de ARM, destacan las porinas y las bombas de eflujo.
Los antibióticos β-lactámicos y algunas fluoroquinolonas que dependen de las porinas para penetrar, se ven afectados debido a mutaciones en dichas proteínas. Además, estas mutaciones pueden surgir durante el tratamiento y, lo que es más importante, potencian la influencia de otros mecanismos de resistencia, como las bombas de eflujo y las enzimas degradativas.
Por otro lado, la expresión de las bombas de eflujo bacterianas, que expulsan los fármacos fuera de la célula, también contribuye en gran medida a la ARM.
- La formación de biofilms bacterianos:
Además de los mecanismos clásicos de resistencia a los antimicrobianos ya mencionados, ahora se reconoce también que el crecimiento dentro de las biopelículas o biofilms puede impedir aún más la actividad antimicrobiana.
Las biopelículas son comunidades microbianas estructuradas, adheridas a la superficie y encerradas en una matriz extracelular que demuestran una gran tolerancia a los agentes antimicrobianos. En particular, las biopelículas desempeñan un papel destacado en las infecciones crónicas.
Se cree que la poca sensibilidad a los antibióticos que presentan las células del biofilm puede variar según la especie y la composición genética del organismo o los organismos, la naturaleza del agente antimicrobiano, la fase de desarrollo de la biopelícula y las condiciones ambientales.
Conclusiones
Aunque la crisis de la salud pública que supone la AMR parece exagerada, es un problema que se lleva hablando y estudiando desde hace tiempo, y ya se han identificado, o se están aplicando, muchos elementos clave para su gestión.
Sin embargo, hasta la fecha, estas medidas no han logrado impedir el avance de la amenaza. Es necesario un enfoque que incorpore a todas las partes interesadas, desde los pacientes y los prescriptores hasta los organismos industriales y gubernamentales. Asimismo, considero que es necesario proporcionar mayor información con respecto a este tema a la población, ya que en muchas ocasiones actuamos de manera incorrecta aún sin saberlo.
Los progresos realizados hasta ahora en la vigilancia del control de las infecciones y de la administración, junto con el descubrimiento y el desarrollo de nuevos antimicrobianos, deben aprovecharse al máximo y perfeccionarse. Sin embargo, se trata de un arduo camino con muchas trabas e inconvenientes. Por ello, es tarea de todos colaborar para intentar frenar el gran aumento de resistencia antimicrobiana que está ocurriendo.
En definitiva, después de décadas de estancamiento, el desarrollo de nuevos antimicrobianos está mostrando los primeros signos de resurgimiento, aunque habrá que ver cómo se manejará la crisis que se está desarrollando, y si realmente «no hay ESKAPE», después de todo.
Artículo editado por Lucía Cejudo.
Bibliografía
– De Oliveira, D. M., Forde, B. M., Kidd, T. J., Harris, P. N., Schembri, M. A., Beatson, S. A., … & Walker, M. J. (2020). Antimicrobial resistance in ESKAPE pathogens. Clinical microbiology reviews, 33(3), e00181-19.
– World Health Organization. 2014. Antimicrobial resistance: global report on surveillance 2014. https://www.who.int/antimicrobial-resistance/ publications/surveillancereport/en/.
Centers for Disease Control and Prevention. 2019. Antibiotic resistant threats in the United States 2019. https://www.cdc.gov/drugresistance/ pdf/threats-report/2019-ar-threats-report-508.pdf.
– Akova M. 2016. Epidemiology of antimicrobial resistance in bloodstream infections. Virulence 7:252–266. https://doi.org/10.1080/21505594.2016 .1159366
-Rice LB. 2008. Federal funding for the study of antimicrobial resistance in nosocomial pathogens: no ESKAPE. J Infect Dis 197:1079–1081. https://doi.org/10.1086/533452.
-World Health Organization. 2017. Global priority list of antibiotic- resistant bacteria to guide research, discovery, and development of new antibiotics. https://www.who.int/medicines/publications/WHO-PPL -Short_Summary_25Feb-ET_NM_WHO.pdf?ua1. Accessed 2 Novem- ber 2019.
–Madigan, M. T., Martinko, J. M., & Parker, J. (2003). Brock. Biología de los microorganismos 12 ed. Editorial Pearson Prentice Hall Iberia, Madrid. Pp, 1064.
-Pendleton, J. N., Gorman, S. P., & Gilmore, B. F. (2013). Clinical relevance of the ESKAPE pathogens. Expert review of anti-infective therapy, 11(3), 297-308.
-Lebreton F, van Schaik W, McGuire AM, Godfrey P, Griggs A, Mazumdar V, Corander J, Cheng L, Saif S, Young S, Zeng Q, Wortman J, Birren B, Willems RJ, Earl AM, Gilmore MS. 2013. Emergence of epidemic multidrug-resistant Enterococcus faecium from animal and commensal strains. mBio 4:e00534-13. https://doi.org/10.1128/mBio.00534-13.
-Murray BE. 1990. The life and times of the Enterococcus. Clin Microbiol Rev 3:46 – 65. https://doi.org/10.1128/cmr.3.1.46.
-Pinholt M, Larner-Svensson H, Littauer P, Moser CE, Pedersen M, Lem- ming LE, Ejlertsen T, Sondergaard TS, Holzknecht BJ, Justesen US, Dzajic E, Olsen SS, Nielsen JB, Worning P, Hammerum AM, Westh H, Jakobsen L. 2015. Multiple hospital outbreaks of vanA Enterococcus faecium in Denmark, 2012–13, investigated by WGS, MLST and PFGE. J Antimicrob Chemother 70:2474 –2482. https://doi.org/10.1093/jac/dkv142.
-Lee T, Pang S, Abraham S, Coombs GW. 2019. Antimicrobial-resistant CC17 Enterococcus faecium: the past, the present and the future. J Glob Antimicrob Resist 16:36–47. https://doi.org/10.1016/j.jgar.2018.08.016.
-Goncalves A, Igrejas G, Radhouani H, Lopez M, Guerra A, Petrucci- Fonseca F, Alcaide E, Zorrilla I, Serra R, Torres C, Poeta P. 2011. Detec- tion of vancomycin-resistant enterococci from faecal samples of Iberian wolf and Iberian lynx, including Enterococcus faecium strains of CC17 and the new singleton ST573. Sci Total Environ 410-411:266–268. https://doi.org/10.1016/j.scitotenv.2011.09.074.
-Gao W, Howden BP, Stinear TP. 2018. Evolution of virulence in Entero- coccus faecium, a hospital-adapted opportunistic pathogen. Curr Opin Microbiol 41:76–82. https://doi.org/10.1016/j.mib.2017.11.030.
-Satilmis L, Vanhems P, Benet T. 2016. Outbreaks of vancomycin- resistant enterococci in hospital settings: a systematic review and calculation of the basic reproductive number. Infect Control Hosp Epidemiol 37:289–294. https://doi.org/10.1017/ice.2015.301.
-Ubeda C, Taur Y, Jenq RR, Equinda MJ, Son T, Samstein M, Viale A, Socci ND, van den Brink MR, Kamboj M, Pamer EG. 2010. Vancomycin-resistant Enterococcus domination of intestinal microbiota is enabled by antibiotic treatment in mice and precedes bloodstream invasion in humans. J Clin Invest 120:4332–4341. https://doi.org/10.1172/JCI43918.
-Silverman JA, Mortin LI, VanPraagh ADG, Li TC, Alder J. 2005. Inhibition of daptomycin by pulmonary surfactant: in vitro modeling and clinical impact. J Infect Dis 191:2149–2152. https://doi.org/10.1086/430352.
-Kourtis AP, Hatfield K, Baggs J, Mu Y, See I, Epson E, Nadle J, Kainer MA, Dumyati G, Petit S, Ray SM, Emerging Infections Program MRSA Author Group, Ham D, Capers C, Ewing H, Coffin N, McDonald LC, Jernigan J, Cardo D, Program EI. 2019. Vital signs: epidemiology and recent trends in methicillin-resistant and in methicillin-susceptible Staphylococcus aureus bloodstream infections—United States. MMWR Morb Mortal Wkly Rep 68:214–219. https://doi.org/10.15585/mmwr.mm6809e1.
-Lim WW, Wu P, Bond HS, Wong JY, Ni KW, Seto WH, Jit M, Cowling BJ. 2019. Determinants of methicillin-resistant Staphylococcus aureus (MRSA) prevalence in the Asia-Pacific region: a systematic review and meta-analysis. J Glob Antimicrob Resist 16:17–27. https://doi.org/10 .1016/j.jgar.2018.08.014.
-Duerden B, Fry C, Johnson AP, Wilcox MH. 2015. The control of methicillin-resistant Staphylococcus aureus blood stream infections in England. Open Forum Infect Dis 2:ofv035. https://doi.org/10.1093/ofid/ ofv035.
-DeLeo FR, Otto M, Kreiswirth BN, Chambers HF. 2010. Community- associated meticillin-resistant Staphylococcus aureus. Lancet 375: 1557–1568. https://doi.org/10.1016/S0140-6736(09)61999-1.
-Dermota U, Jurca T, Harlander T, Kosir M, Zajc U, Golob M, Zdovc I, Kosnik IG. 2016. Infections caused by community-associated methicillin-resistant Staphylococcus aureus European clone (St80) in Slovenia between 2006 and 2013. Zdr Varst 55:121–125. https://doi .org/10.1515/sjph-2016-0018.
-van Alen S, Ballhausen B, Peters G, Friedrich AW, Mellmann A, Kock R, Becker K. 2017. In the centre of an epidemic: fifteen years of LA-MRSA CC398 at the university hospital Munster. Vet Microbiol 200:19–24. https://doi.org/10.1016/j.vetmic.2016.01.021.
-Hryniewicz MM, Garbacz K. 2017. Borderline oxacillin-resistant Staphy- lococcus aureus (BORSA)—a more common problem than expected? J Med Microbiol 66:1367–1373. https://doi.org/10.1099/jmm.0.000585.
-Cosgrove SE, Sakoulas G, Perencevich EN, Schwaber MJ, Karchmer AW, Carmeli Y. 2003. Comparison of mortality associated with methicillin- resistant and methicillin-susceptible Staphylococcus aureus bacteremia: a meta-analysis. Clin Infect Dis 36:53–59. https://doi.org/10.1086/345476.
-Tzouvelekis LS, Markogiannakis A, Psichogiou M, Tassios PT, Daikos GL. 2012. Carbapenemases in Klebsiella pneumoniae and other Enterobacteriaceae: an evolving crisis of global dimensions. Clin Micro- biol Rev 25:682–707. https://doi.org/10.1128/CMR.05035-11.
-David S, Reuter S, Harris SR, Glasner C, Feltwell T, Argimon S, Abudahab K, Goater R, Giani T, Errico G, Aspbury M, Sjunnebo S, EuSCAPE Working Group, ESGEM Study Group, Feil EJ, Rossolini GM, Aanensen DM, Grundmann H. 2019. Epidemic of carbapenem-resistant Klebsiella pneu- moniae in Europe is driven by nosocomial spread. Nat Microbiol 4:1919 –1929. https://doi.org/10.1038/s41564-019-0492-8.
-Wei DD, Wan LG, Deng Q, Liu Y. 2016. Emergence of KPC-producing Klebsiella pneumoniae hypervirulent clone of capsular serotype K1 that belongs to sequence type 11 in mainland China. Diagn Microbiol Infect Dis 85:192–194. https://doi.org/10.1016/j.diagmicrobio.2015.03.012.
-Munoz-Price LS, Weinstein RA. 2008. Acinetobacter infection. N Engl J Med 358:1271–1281. https://doi.org/10.1056/NEJMra070741.
-Villegas MV, Hartstein AI. 2003. Acinetobacter outbreaks, 1977–2000. Infect Control Hosp Epidemiol 24:284 –295. https://doi.org/10.1086/ 502205.
-Lob SH, Hoban DJ, Sahm DF, Badal RE. 2016. Regional differences and trends in antimicrobial susceptibility of Acinetobacter baumannii. Int J Antimicrob Agents 47:317–323. https://doi.org/10.1016/j .ijantimicag.2016.01.015.
-Zarrilli R, Pournaras S, Giannouli M, Tsakris A. 2013. Global evolution of multidrug-resistant Acinetobacter baumannii clonal lineages. Int J An- timicrob Agents 41:11–19. https://doi.org/10.1016/j.ijantimicag.2012.09 .008.
-Gellatly SL, Hancock R. 2013. Pseudomonas aeruginosa: new insights into pathogenesis and host defenses. Pathog Dis 67:159–173. https:// doi.org/10.1111/2049-632X.12033.
-Treepong P, Kos VN, Guyeux C, Blanc DS, Bertrand X, Valot B, Hocquet D. 2018. Global emergence of the widespread Pseudomonas aeruginosa ST235 clone. Clin Microbiol Infect 24:258 –266. https://doi.org/10.1016/ j.cmi.2017.06.018.
-Cabot G, Lopez-Causape C, Ocampo-Sosa AA, Sommer LM, Dominguez MA, Zamorano L, Juan C, Tubau F, Rodriguez C, Moya B, Pena C, Martinez-Martinez L, Plesiat P, Oliver A, Spanish Network for Research in Infectious Diseases. 2016. Deciphering the resistome of the wide- spread Pseudomonas aeruginosa sequence type 175 international high- risk clone through whole-genome sequencing. Antimicrob Agents Che- mother 60:7415–7423. https://doi.org/10.1128/AAC.01720-16.
-Newman JW, Floyd RV, Fothergill JL. 2017. The contribution of Pseudomonas aeruginosa virulence factors and host factors in the establishment of urinary tract infections. FEMS Microbiol Lett 364:fnx124. https://doi.org/10.1093/femsle/fnx124.
-Hoiby N, Bjarnsholt T, Givskov M, Molin S, Ciofu O. 2010. Antibiotic resistance of bacterial biofilms. Int J Antimicrob Agents 35:322–332. https://doi.org/10.1016/j.ijantimicag.2009.12.011.
-Davin-Regli A, Pagès J-M. 2015. Enterobacter aerogenes and Enterobacter cloacae; versatile bacterial pathogens confronting antibiotic treatment. Front Microbiol 6:392. https://doi.org/10.3389/fmicb.2015.00392.
-Chevalier J, Mulfinger C, Garnotel E, Nicolas P, Davin-Régli A, Pagès J-M. 2008. Identification and evolution of drug efflux pump in clinical Enterobacter aerogenes strains isolated in 1995 and 2003. PLoS One 3:e3203. https://doi.org/10.1371/journal.pone.0003203.
-Malek A, McGlynn K, Taffner S, Fine L, Tesini B, Wang J, Mostafa H, Petry S, Perkins A, Graman P, Hardy D, Pecora N. 2019. Next-generation sequencing based hospital outbreak investigation yields insight into Klebsiella aerogenes population structure and determinants of carbapenem resistance and pathogenicity. Antimicrob Agents Chemother 63:e02577-18. https://doi.org/10.1128/AAC.02577-18.
-Thiolas A, Bollet C, La Scola B, Raoult D, Pagès J-M. 2005. Successive emergence of Enterobacter aerogenes strains resistant to imipenem and colistin in a patient. Antimicrob Agents Chemother 49:1354–1358. https://doi.org/10.1128/AAC.49.4.1354-1358.2005.
-Band VI, Crispell EK, Napier BA, Herrera CM, Tharp GK, Vavikolanu K, Pohl J, Read TD, Bosinger SE, Trent MS, Burd EM, Weiss DS. 2016. Antibiotic failure mediated by a resistant subpopulation in Enterobacter cloacae. Nat Microbiol 1:16053. https://doi.org/10.1038/ nmicrobiol.2016.53.
-Naas T, Oueslati S, Bonnin RA, Dabos ML, Zavala A, Dortet L, Retailleau P, Iorga BI. 2017. Beta-Lactamase Database (BLDB—structure and function. J Enzyme Inhib Med Chem 32:917–919. https://doi.org/10.1080/ 14756366.2017.1344235.
-Shaw KJ, Rather PN, Hare RS, Miller GH. 1993. Molecular genetics of aminoglycoside resistance genes and familial relationships of the aminoglycoside-modifying enzymes. Microbiol Rev 57:138 –163.
-Lakhundi S, Zhang K. 2018. Methicillin-resistant Staphylococcus aureus: molecular characterization, evolution, and epidemiology. Clin Microbiol Rev 31:e00020-18. https://doi.org/10.1128/CMR.00020-18.
-Hasdemir UO, Chevalier J, Nordmann P, Pages JM. 2004. Detection and prevalence of active drug efflux mechanism in various multidrug- resistant Klebsiella pneumoniae strains from Turkey. J Clin Microbiol 42:2701–2706. https://doi.org/10.1128/JCM.42.6.2701-2706.2004.
-Pages JM, James CE, Winterhalter M. 2008. The porin and the permeating antibiotic: a selective diffusion barrier in Gram-negative bacteria. Nat Rev Microbiol 6:893–903. https://doi.org/10.1038/nrmicro1994.
-Li XZ, Plesiat P, Nikaido H. 2015. The challenge of efflux-mediated antibiotic resistance in Gram-negative bacteria. Clin Microbiol Rev 28:337–418. https://doi.org/10.1128/CMR.00117-14.
-Hill D, Rose B, Pajkos A, Robinson M, Bye P, Bell S, Elkins M, Thompson B, MacLeod C, Aaron SD, Harbour C. 2005. Antibiotic susceptibilities of Pseudomonas aeruginosa isolates derived from patients with cystic fibrosis under aerobic, anaerobic, and biofilm conditions. J Clin Microbiol 43:5085–5090. https://doi.org/10.1128/JCM.43.10.5085-5090.2005.
-Nickel JC, Ruseska I, Wright JB, Costerton JW. 1985. Tobramycin resistance of Pseudomonas aeruginosa cells growing as a biofilm on urinary catheter material. Antimicrob Agents Chemother 27:619 – 624. https:// doi.org/10.1128/aac.27.4.619.
-Percival SL, Suleman L, Vuotto C, Donelli G. 2015. Healthcare-associated infections, medical devices and biofilms: risk, tolerance and control. J Med Microbiol 64:323–334. https://doi.org/10.1099/jmm.0.000032.
-Rice LB. Federal funding for the study of antimicrobial resistance in nosocomial pathogens: no ESKAPE. J. Infect. Dis. 197(8), 1079–1081 (2008).
-Boucher HW, Talbot GH, Bradley JS et al. Bad bugs, no drugs: no ESKAPE! An update from the Infectious Diseases Society of America. Clin. Infect. Dis. 48(1), 1–12 (2009).
-Elsner H, Sobottka I, Mack D, Laufs R, Claussen M, Wirth R. Virulence factors of Enterococcus faecalis and Enterococcus faecium blood culture isolates. Eur. J. Clin. Microbiol. Infect. Dis. 19(1), 39–42 (2000).
-Van Wamel WJ, Hendrickx AP, Bonten MJ, Top J, Posthuma G, Willems RJ. Growth condition-dependent Esp expression by Enterococcus faecium affects initial adherence and biofilm formation. Infect. Immun. 75(2), 924–931 (2007).
-Costerton JW, Stewart PS, Greenberg EP. Bacterial biofilms: a common cause of persistent infections. Science 284(5418), 1318–1322 (1999).
-Chambers HF, Deleo FR. Waves of resistance: Staphylococcus aureus in the antibiotic era. Nat. Rev. Microbiol. 7(9), 629–641 (2009).
-Bush K. Alarming β-lactamase-mediated resistance in multidrug-resistant Enterobacteriaceae. Curr. Opin. Microbiol. 13(5), 558–564 (2010).
-Brink AJ, Coetzee J, Clay CG et al. Emergence of New Delhi metallo-β- lactamase (NDM-1) and Klebsiella pneumoniae carbapenemase (KPC-2) in South Africa. J. Clin. Microbiol. 50(2), 525–527 (2012).
-Biendo M, Laurans G, Lefebvre JF, Daoudi F, Eb F. Epidemiological study of an Acinetobacter baumannii outbreak by using a combination of antibiotyping and ribotyping. J. Clin. Microbiol. 37(7), 2170–2175 (1999).
-Vila J, Martí S, Sánchez-Céspedes J. Porins, efflux pumps and multidrug resistance in Acinetobacter baumannii. J. Antimicrob. Chemother. 59(6), 1210–1215 (2007).
-Livermore DM. Multiple mechanisms of antimicrobial resistance in Pseudomonas aeruginosa: our worst nightmare? Clin. Infect. Dis. 34(5), 634–640 (2002).
-Castanheira M, Deshpande LM, Mathai D, Bell JM, Jones RN, Mendes RE. Early dissemination of NDM-1- and OXA- 181-producing Enterobacteriaceae in Indian hospitals: report from the SENTRY Antimicrobial Surveillance Program, 2006–2007. Antimicrob. Agents Chemother. 55(3), 1274–1278 (2011).
-Butler MS, Cooper MA. Antibiotics in the clinical pipeline in 2011. J. Antibiot. 64(6), 413–425 (2011).





